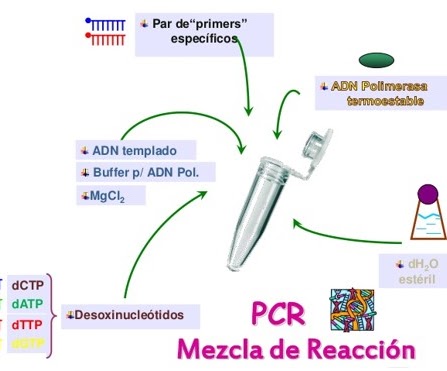
Amplificación de secuencias barcode
Preparar una mezcla de reactivos para detectar y copiar la secuencia seleccionada como diana en el ADN de los microroganismos:
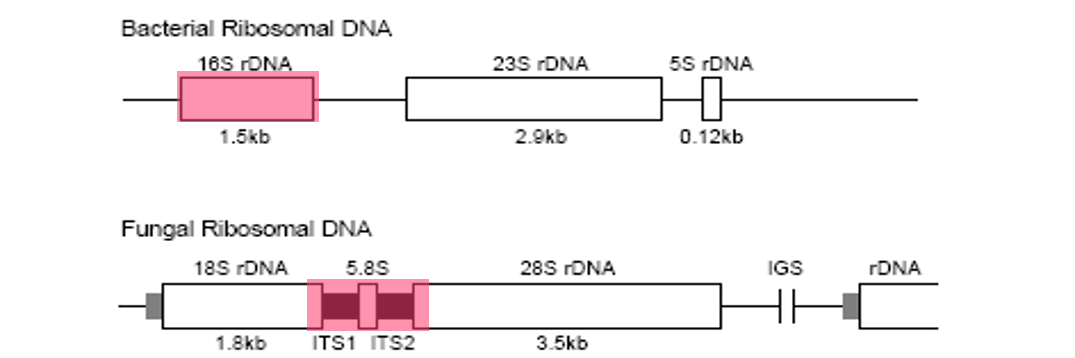
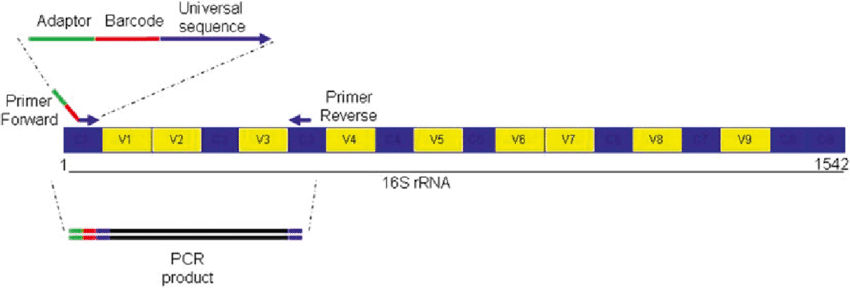
– en el caso de las bacterias la diana será un fragmento del ADN que codifica para la subunidad pequeña del ribosoma (16S)
– en el caso de las levaduras, un fragmento que incluye las regiones espaciadoras (ITS) que hay entre el gen que codifica para la subunidad pequeña 18S y la subunidad grande 28S, que a su vez incluye la pequeña subunidad 5,8S.
En esta mezcla será sólo diferentes en los cebadores (primers) que son los que deben unirse específicamente a las regiones diana. Los demás elementos son iguales. Para hacer la mezcla calculamos sobre la base de un volumen de reacción de 25 μL para cada muestra, con lo que para N muestras la composición será la que aparece en la figura.
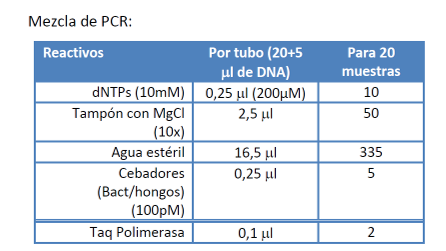
Mezclar suavemente los componentes de la reacción y repartir 20 microlitros en los tubos de PCR.
Añadir a cada muestra 5 microlitros del DNA extraído teniendo en cuenta si es DNA bacteriano o fungico para amplificarlo con los primers correspondientes.
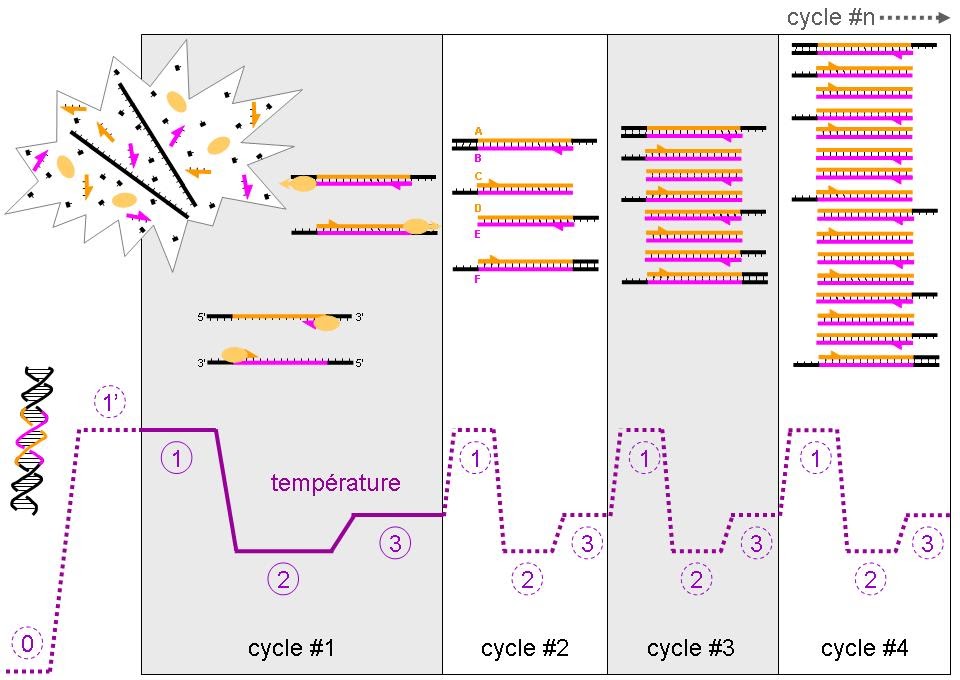
Llevar al termociclador con el siguiente programa:
1 ciclo: 95ºC ………….5 min
35 ciclos: 95ºC………….30 sg. / 55ºC……30 sg. / 72ºC ….. 1 min
1 ciclo: 72ºC ,,,,,,,6 min
Apoyo a acciones de Innovación Docente – Vicerrectorado de Estudios / Consultas e incidencias técnicas- Tlf: 96 522 2059 –